Zellmembranen super aufgelöst
02.12.2020Mit der Expansionsmikroskopie lassen sich erstmals auch feinste Details von Zellmembranen abbilden. Das bietet neue Einblicke in bakterielle und virale Infektionsprozesse.
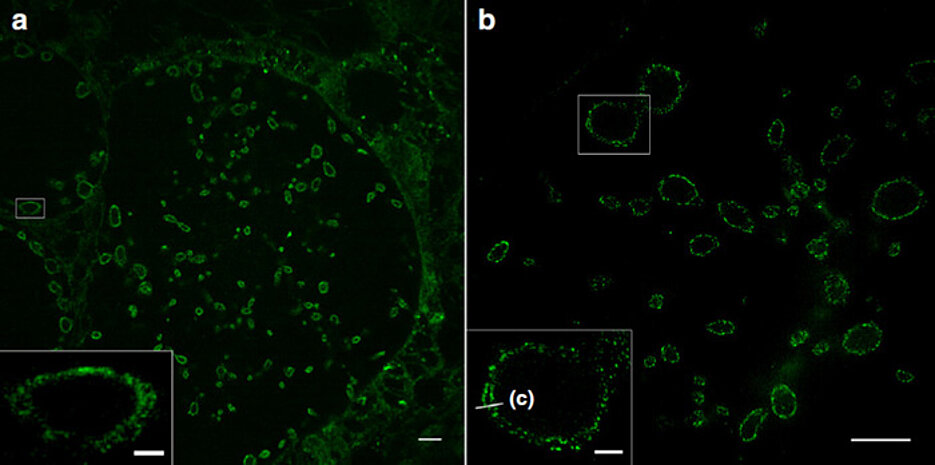
Mit der Expansionsmikroskopie (ExM) lassen sich Zellen und ihre Bausteine mit einer räumlichen Auflösung weit unterhalb von 200 Nanometern abbilden. Dazu werden die Proteine der zu untersuchenden Probe in ein schwellbares Polymer vernetzt. Nach der Zerstörung der Wechselwirkungen zwischen den Molekülen können die Proben mit Wasser um ein Vielfaches ausgedehnt werden. Das macht detaillierte Einblicke in ihre Strukturen möglich.
„Diese Methode war bisher auf Proteine beschränkt. Im Journal Nature Communications stellen wir jetzt eine Möglichkeit vor, wie wir auch Lipide und damit Zellmembranen expandieren können“, sagt Professor Markus Sauer, Experte für hochauflösende Mikroskopie vom Biozentrum der Julius-Maximilians-Universität Würzburg (JMU). Beteiligt an der Publikation sind auch die JMU-Professoren Thomas Rudel (Mikrobiologie) und Jürgen Seibel (Chemie).
Synthetische Lipide werden markiert und expandiert
Das Team von Jürgen Seibel hat funktionalisierte Sphingolipide synthetisiert, die ein wichtiger Bestandteil von Zellmembranen sind. Gibt man diese Lipide zu Zellkulturen, werden sie in die Zellmembranen eingebaut. Im Anschluss lassen sie sich mit einem Farbstoff markieren und in einem schwellbaren Polymer vier- bis zehnfach expandieren.
Die JMU-Forscher zeigen, dass sich auf diese Weise – in Kombination mit der Strukturierten Beleuchtungsmikroskopie (SIM) – erstmals verschiedene Membranen und ihre Wechselwirkungen mit den Zellproteinen mit einer Auflösung von 10 bis 20 Nanometern abbilden lassen: Zellmembranen, die äußere und die innere Zellkernmembran und auch die Membranen intrazellulärer Organellen wie Mitochondrien.
Membranen von Bakterien und Viren im Blick
Die Sphingolipide bauen sich außerdem hocheffizient in die Membranen von Bakterien ein. Damit können nun erstmals Krankheitserreger wie Neisseria gonorrhoeae, Chlamydia trachomatis und Simkania negevensis in infizierten Zellen in einer Auflösung dargestellt werden, die bisher nur mit der Elektronenmikroskopie erreicht wurde. Sogar die innere und die äußere Membran von gramnegativen Bakterien lassen sich voneinander unterscheiden.
„Mit den neuen superauflösenden mikroskopischen Verfahren wollen wir jetzt bakterielle Infektionsmechanismen und Ursachen für Antibiotikaresistenzen untersuchen. Was wir dabei lernen, lässt sich womöglich für verbesserte Therapien nutzen“, sagt Professor Thomas Rudel, Experte für bakterielle Infektionen.
Womöglich integrieren sich die Sphingolipide auch in die Membran von Viren. Falls das gelingt, könnte man die Wechselwirkungen von Coronaviren mit Zellen erstmals lichtmikroskopisch in hoher Auflösung untersuchen.
Die beschriebenen Arbeiten wurden von der Deutschen Forschungsgemeinschaft (DFG) im Rahmen der Graduiertenkollegs 2157 und 2581 finanziell gefördert.
Publikation
Nanoscale imaging of bacterial infections by sphingolipid expansion microscopy. Nature Communications, 2. Dezember 2020, DOI: 10.1038/s41467-020-19897-1
Kontakt
Prof. Dr. Thomas Rudel, Lehrstuhl für Mikrobiologie, Universität Würzburg,
T -49 931 31-84401, thomas.rudel@biozentrum.uni-wuerzburg.de
Prof. Dr. Markus Sauer, Lehrstuhl für Biotechnologie und Biophysik, Universität Würzburg,
T +49 931 31-88687, m.sauer@uni-wuerzburg.de